Wykupienie dostępu pozwoli Ci czytać artykuły wysokiej jakości i wspierać niezależne dziennikarstwo w wymagających dla wydawców czasach. Rośnij z nami! Pełna oferta →
Kiedy Nikolai Peristov w 2008 r. spacerował brzegiem rzeki Irtysz w okręgu Ust’-Ishim na środkowej Syberii, nie spodziewał się, jak cenne okaże się jego znalezisko. Nikolai jest artystą trudniącym się rzeźbieniem biżuterii w mamucich kłach. Jednak kość, którą przypadkowo zauważył tego dnia, nie należała do mamuta.
Policyjni specjaliści ocenili, że to bardzo stara kość udowa człowieka. Jej wiek zmierzono w Instytucie Antropologii Ewolucyjnej Maxa Plancka w Lipsku za pomocą datowania izotopowego: wynosi od 43 do 47 tysięcy lat. Potem kość trafiła w ręce profesora Svantego Pӓӓbo – archeologa genomu.
Jego zespół wyizolował i oczyścił DNA z kości, a następnie poddał sekwencjonowaniu i analizie. W ten sposób poznaliśmy kompletną sekwencję genomu najstarszego człowieka, którego szczątki badano do tej pory. Wyniki badań ukazały się w jednym z październikowych wydań „Nature”.
Genetyczne kalendarium
Svante Pӓӓbo jest dziś dyrektorem wydziału genetyki we wspomnianym Instytucie Maxa Plancka. Zaczął studiować egiptologię – ale się rozczarował: zamiast podróżować w poszukiwaniu nowych stanowisk badawczych, spędzał dni na odmianie starożytnych egipskich czasowników. Śladami ojca – Sunego Karla Bergströma, laureata Nobla z fizjologii i medycyny w 1982 r. – poszedł więc na medycynę i podjął studia doktoranckie z immunologii molekularnej. Jednak mumie wciąż nie dawały mu spokoju.
Starzy znajomi z egiptologii dostarczyli Svantemu fragmenty skóry i kości mumii. Wieczorami i w weekendy pracował nad izolacją materiału genetycznego z tych próbek. Udało się. Zanalizował krótki fragment DNA sprzed ponad 2 tys. lat. Wyniki wysłał do „Nature”. To był strzał w dziesiątkę. Od tej pory nazwisko Pӓӓbo będzie się kojarzyło z przełomowymi odkryciami w archeologii genomu, zwanej też paleogenetyką.
O tym, że DNA jest nośnikiem informacji genetycznej, wiadomo od lat 40. XX w. Ale dopiero niedawno naukowcy zorientowali się, że można patrzeć na DNA również jak na historyczne kalendarium. Dziedziczymy je od naszych rodziców, którzy z kolei odziedziczyli je od naszych dziadków, ci od pradziadków itd. DNA, jak to ujęto na łamach „Smithsonian Magazine”, jest jak „starożytny manuskrypt przepisywany z pokolenia na pokolenie, w którym zawarte są opowieści wykraczające poza naszą pamięć”.
Genom to długie cząsteczki DNA zorganizowane w chromosomy, obecne we wnętrzu prawie każdej komórki. Informacja genetyczna zapisana jest w nich za pomocą kodu czterech związków organicznych, oznaczanych literami A, G, T i C. Przy każdym podziale komórki DNA musi zostać powielone. Podczas kopiowania zdarzają się błędy i np. zamiast G w łańcuch wbudowane zostaje A. Takie mutacje, gdy nie wykryją ich mechanizmy naprawcze, są przekazywane kolejnym generacjom komórek. A jeżeli mają miejsce w DNA komórek rozrodczych – kolejnym pokoleniom. Zmiany te, zwane SNP (ang. single nucleotide polymorphism), które nie wywołują chorób genetycznych, często znajdują się w częściach genomu o nieznanych jeszcze funkcjach. Naukowcy mogą je śledzić, porównując ludzi między sobą, ale też z innymi gatunkami.
Odrobina neandertalczyka
Cząsteczka DNA zawiera informację o życiu, musi więc być niezwykle odporna. Można ją podgrzać do stu stopni Celsjusza albo schłodzić do temperatury ciekłego azotu, przetrwa też wiele tysięcy lat w kościach zagrzebanych w ziemi. „DNA odzyskane ze szczątków archeologicznych lub paleontologicznych pozwala cofnąć się w czasie i badać genetyczne zależności pomiędzy wymarłymi organizmami i ich współczesnymi krewnymi – pisze prof. Pӓӓbo w jednym z artykułów. – To stwarza całkiem nową perspektywę spojrzenia na ewolucję”.
Kluczowa w paleogenetyce jest izolacja materiału genetycznego z kości. Należy starannie oddzielić DNA pochodzące od bakterii i grzybów, których w szczątkach jest bardzo dużo, a ponadto trzeba pracować tak, by nie zanieczyścić próbki własnym DNA. Wygląda to podobnie jak w laboratoriach kryminalistycznych. Wyizolowane krótkie fragmenty DNA są następnie powielane, żeby można je było poddać analizie. Dziś etap odczytania sekwencji A, G, C i T to najprostsza część procedury. Ponieważ DNA jest bardzo pokawałkowane, wynikiem sekwencjonowania są miliony krótkich odczytów, które trzeba ułożyć w całość, czyli genom. To praca dla zastępu bioinformatyków. Ten etap trwa najdłużej, wymaga godzin pracy i wielkiej mocy obliczeniowej komputerów. Otrzymana kompletna sekwencja genomu jest następnie porównywana do uśrednionej sekwencji DNA człowieka i jego małpiego przodka – szympansa.
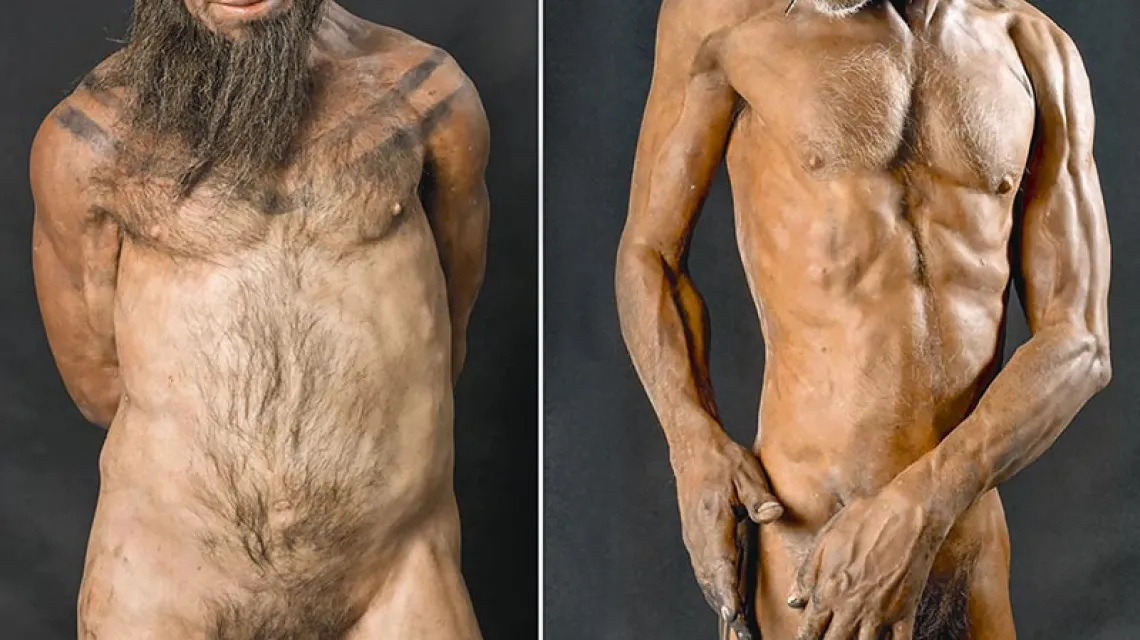
Naukowców od 150 lat szczególnie interesują losy neandertalczyków – ludu zamieszkującego Europę i część Azji około 300 tys. lat temu. Co się stało, kiedy spotkali się z Homo sapiens, który opuścił Afrykę? Czy doszło do krzyżowań między ludźmi a neandertalczykami? Dlaczego jedni podróżują teraz w kosmos, a drudzy wymarli z nieznanych powodów tysiące lat temu? Czy paleogenetyka może odpowiedzieć na te pytania? Prof. Pӓӓbo szuka odpowiedzi w DNA.
W 2010 r. „Science” opublikowało zarys genomu neandertalczyka, a na początku tego roku zaprezentowano już całkowity zapis sekwencji jego DNA. Z analiz wynika, że ludzie spotkali neandertalczyków na Bliskim Wschodzie, zaraz po wyjściu z Afryki. Tam doszło do zmieszania dwóch genomów. Dzisiaj w potomkach ludów, które opuściły Afrykę (czyli również w nas) żyje odrobina neandertalczyka. Dokładnie jest to 2,5 proc. ich całkowitego genomu. W genach kodujących białka w ludzkim genomie – a jest ich ok. 20 tys. – jest tylko 96 zmian w porównaniu do neandertalczyka. Różnice wykazano m.in. w genach odpowiedzialnych za rozwój mowy i mózgu. Czy to właśnie te zmiany uczyniły nas współczesnymi ludźmi i zapewniły sukces ewolucyjny?
Puzzle z pradziejów
Kogo jeszcze spotkali Homo sapiens w wędrówce przez kontynent azjatycki? Najnowsze badania laboratoriów w Lipsku wskazują na lud denisowian. Jak w przypadku neandertalczyka, którego miano pochodzi od doliny Neandertal w Niemczech, tak i tu nazwa wzięła się od stanowiska archeologicznego, gdzie odkryto szczątki. Jaskinia Denisowa znajduje się w górach Ałtaj na Syberii. Znaleziono tam pojedynczą kość paliczka i dwa zęby, z których udało się wyizolować DNA. Na razie wiemy o tym ludzie niewiele. Genetycznie denisowianie są podobni do neandertalczyków, a ich cząstka również żyje współcześnie. Ich ślady genetyczne utrwaliły się na poziomie około 5 proc. w genomach dzisiejszych mieszkańców wysp Oceanu Spokojnego i Papui-Nowej Gwinei. Nie wiadomo, ile jeszcze takich puzzli czeka w różnych regionach świata.
Celem Svantego Pӓӓbo jest, jak mówi, „identyfikacja zmian genetycznych, które zrobiły z nas ludzi”. Naukowcy w końcu będą mogli określić, które geny uległy zmianom, kiedy się zmieniły, a nawet dlaczego doszło do konkretnych modyfikacji. Kilka lat temu opinię publiczną zainteresował gen FOXP2, okrzyknięty przez media „genem mowy”. U ludzi doszło w nim do modyfikacji, która mogła się przyczynić do wykształcenia się mowy: osoby z inną mutacją FOXP2 mają problemy z nauką mówienia. Myszy z wprowadzoną do genomu ludzką wersją tego genu szybciej uczą się wykonywania pewnych zadań. Ale w genetyce nic nie jest tak proste, za rozwój mowy musi odpowiadać wiele różnych genów: bo żadna mysz nie przemówiła ludzkim głosem.
ANNA BARTOSIK jest doktorem nauk biologicznych Uniwersytetu w Heidelbergu. Pracuje w Międzynarodowym Instytucie Biologii Molekularnej i Komórkowej w Warszawie.
MITOCHONDRIALNA EWA
Materiał genetyczny budujący chromosomy to nie jedyne cząsteczki DNA w naszych komórkach. W mitochondriach – komórkowych elektrowniach – obecne są koliste cząsteczki DNA. Ta część informacji genetycznej dziedziczona jest zawsze od matki – mitochondria z plemnika nie trafiają do zapłodnionej komórki jajowej, która ma własne. Tylko kobiety, które mają córki, przekazują mitochondrialne DNA (mtDNA). Ponieważ średnio raz na tysiąc pokoleń zdarzają się w nim punktowe mutacje, śledzenie ich zmienności w próbkach od różnych osób pozwala odtworzyć wygląd mtDNA wspólnego przodka wszystkich ludzi w linii żeńskiej, nazwanego „mitochondrialną Ewą”, a także przybliżony czas jej występowania. „Ewa” nie musiała być jedną kobietą – była to populacja kobiet o takim samym mtDNA, które przekazały dalej.
Y-CHROMOSOMALNY ADAM
W trakcie podziału komórkowego chromosomy od matki i od ojca wymieniają między sobą fragmenty DNA. Proces ten, nazywany po angielsku crossing-over, jest kluczowy dla utrzymania zmienności genetycznej w populacji. Do takich wymian dochodzi jedynie między chromosomami, które są komplementarne, tzn. kodują te same geny. Tylko chromosom Y w męskich komórkach nie ma swojego odpowiednika. A zatem jest przekazywany z ojca na syna w takiej samej formie przez pokolenia. Dlatego, analizując punktowe mutacje DNA chromosomu Y, można dotrzeć do wspólnego przodka Homo sapiens w linii męskiej: „y-chromosomalnego Adama”.